博客
攻略:TCGA三级数据下载技巧
作为目前最大的癌症基因信息的数据库,TCGA可能蕴藏着难以想象的宝贵信息,围绕TCGA已经有很多重量级文章出现,但 TCGA数据库并入GDC以后,以前的有些链接失效,导致很多战友不知道如何下载TCGA三级数据。有些战友怀疑老美故意借此机会坑害中国科研人员,其实是不可能的,仔细想想这个账划不来。我坚信有链接能够找到三级数据,花了两个小时,找到了链接,还发现这种方法比assembler下载更方便,能用上迅雷。
网速可以得话,三个小时,能够下载所有的TCGA三级数据,结果就是这么任性!其实任何生物信息的网站使用都比较简单,大家耐心的多看看help,多摸索几次,还是很容易上手的。
点击 launch data portal
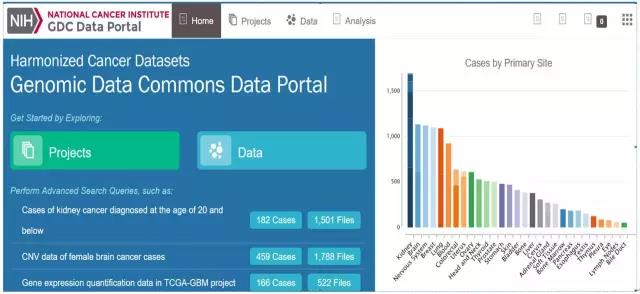
点击 Analysis

点击Broad Firehose,
大家注意这个页面中在firehose的上面一个按钮,可以进入熟悉的在线分析界面(cBioPortal);
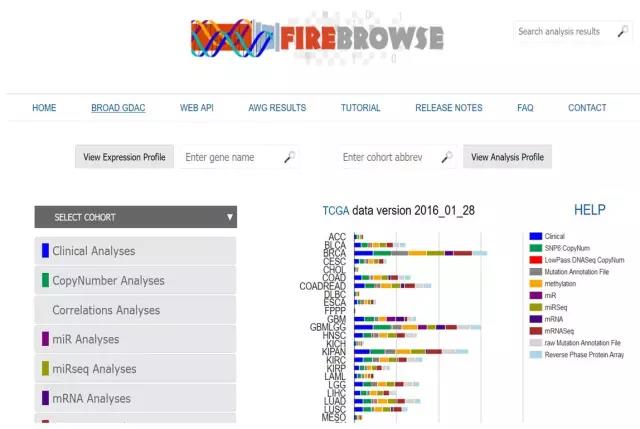
这个界面可以清楚地看到数据版本是2016年1月28日;
在select cohort下面有很多分析内容,可以直接分析,但我没尝试过。在这一列的右边,我们可以看到很多肿瘤的相关数据,都是三级数据。我们选择宫颈癌为例(CESC)为例下载数据。
点击SELECT COHORT,这时候会出现很多肿瘤的下拉列表,在里面选择你想要的癌症类型(我们选择宫颈癌):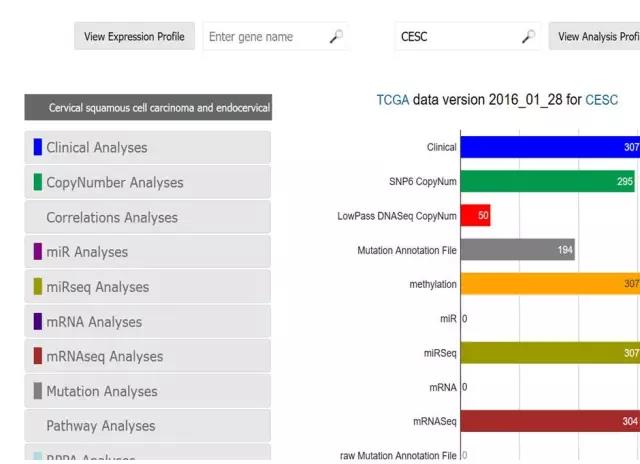
这时候,我们看到很多宫颈癌的数据,307例临床数据,295例SNP数据,还有甲基化,突变,miRseq,mRNAseq的数据等等。怎么下载了?
非常容易,想下载什么数据,就点击什么数据,例如我们下载临床数据,就点击clinical后面的深蓝色条条,弹出的窗口是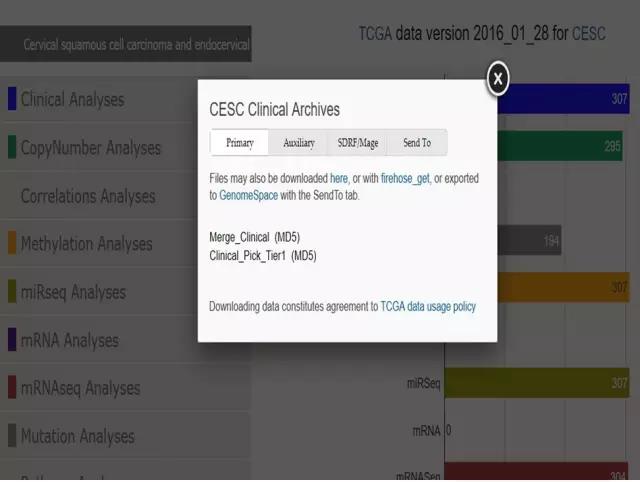
临床数据已经出来了,想下载那个,就选择哪个。我选择合并的数据。点击merge-clinical即可,记住右键点击可以迅雷下载!
我们选择mRNAseq数据下载,点击后面的红色条条,出来的结果是这样的:

结果就出来了,选择自己需要的哪一个就行了。这是我常用的数据类型
illuminahiseq_rnaseqv2-RSEM_genes (MD5)
至于为什么这个数据类型,其实我不是很懂,我用assembler下载数据的时候,也是用的这个数据类型。这里面有原始数据,和归一化的数据。
